HOG(1)ではOpenCVだったので手抜きです。 次はスクラッチしてみたものです。
#include <iostream>
#include <string>
#include <fstream>
#include <vector>
#include <opencv/cv.h>
#include <opencv2/core.hpp>
#include <opencv2/imgcodecs.hpp>
#include <opencv2/imgproc.hpp>
#include <opencv2/highgui.hpp>
#define N_BIN 9 // ヒストグラムのビン数
#define THETA (180 / N_BIN) // 分解能
#define CELL_SIZE 20 //セルサイズ (pixel)
#define BLOCK_SIZE 3 //ブロックサイズ (セル)
#define PI 3.14
/*
* HOG 特徴量の書き出し
*/
int OutputHOGFeature(int nFeatureNum, double *pHOGFeatures, char* FileName)
{
char buf[256];
FILE *fp;
sscanf(FileName, "%s.png", buf);
strcat(buf, ".hog");
if(NULL == (fp=fopen(buf, "w"))){
printf("Can not open the outfile.\n");
return(1);
}
for(int i=0; i<nFeatureNum; i++){
fprintf(fp, "%lf\n", pHOGFeatures[i]);
}
fclose(fp);
return(0);
}
/*
* 勾配方向ヒストグラムの算出
*/
int CompHistogram(cv::Mat Image, double cell_hist[])
{
int num1, num2; //配列の要素数の計算
int xDelta,yDelta; //各方向の輝度の差分
double magnitude; //勾配強度
double angle = 180.0/(double)N_BIN; //量子化する時の角度
double gradient;
unsigned char* imgSource = (unsigned char*)&Image.data[0];
// パッチ内の移動
for(int y=0; y<Image.rows; y++){
if(Image.rows/CELL_SIZE <= y/CELL_SIZE) continue;
for(int x=0; x<Image.cols; x++){
if(Image.cols/CELL_SIZE <= x/CELL_SIZE) continue;
// 横方向の差分
if(x == 0){
num1 = y*Image.cols+(x+0);
num2 = y*Image.cols+(x+1);
}else if(x == Image.cols-1){
num1 = y*Image.cols+(x-1);
num2 = y*Image.cols+(x+0);
}else{
num1 = y*Image.cols+(x-1);
num2 = y*Image.cols+(x+1);
}
xDelta = imgSource[num1]-imgSource[num2];
// 縦方向の差分
if(y == 0){
num1 = (y+0)*Image.cols+x;
num2 = (y+1)*Image.cols+x;
}else if(y == Image.rows-1){
num1 = (y-1)*Image.cols+x;
num2 = (y+0)*Image.cols+x;
}else{
num1 = (y-1)*Image.cols+x;
num2 = (y+1)*Image.cols+x;
}
yDelta = imgSource[num1]-imgSource[num2];
// 勾配強度の算出
magnitude = sqrt((double)xDelta*(double)xDelta+(double)yDelta*(double)yDelta);
gradient = atan2((double)yDelta, (double)xDelta); // 勾配方向の算出
gradient = (gradient*180.0)/PI; // ラジアンから角度へ変換
if(gradient < 0.0) gradient += 360.0; // 符号が負である場合は反転
if(gradient > 180.0) gradient -= 180.0; // 0~360度から 0~180度に変換
gradient = gradient/angle; // 20度ずつ,9分割
// ヒストグラムに蓄積
num1 = (int)((y/CELL_SIZE)*(Image.cols/CELL_SIZE)*N_BIN+(x/CELL_SIZE)*N_BIN+(int)gradient);
cell_hist[num1] += magnitude;
}
}
return(0);
}
/*
* HOG 特徴量の算出と正規化
*/
int CompHOG(double *pHOGFeatures, double cell_hist[], int Cell_Y, int Cell_X)
{
int x,y,i,j,k;
int num;
double sum_magnitude;
// 特徴量の算出と正規化
for(y=0; y<Cell_Y; y++){
for(x=0; x<Cell_X; x++){
// 勾配方向¸
sum_magnitude = 0.0;
for(k=0; k<N_BIN; k++){
// 特徴量量を正規化する時に使用する特徴量の二乗和を正規化
for(j=-((int)(BLOCK_SIZE/2)); j<=(int)(BLOCK_SIZE/2); j++){
if(((y+j) < 0) || ((y+j) >= Cell_Y)) continue;
for(i=-((int)(BLOCK_SIZE/2)); i<=(int)(BLOCK_SIZE/2); i++){
if(((x+i) < 0) || ((x+i) >= Cell_X)) continue;
// 正規化のためヒストグラムの総和の二乗を算出
num = (int)((y+j)*Cell_X*N_BIN+(x+i)*N_BIN+k);
sum_magnitude += cell_hist[num] * cell_hist[num];
}
}
}
sum_magnitude = sqrt(sum_magnitude);
for(k=0; k<N_BIN; k++){
// 特徴量の正規化
num = (int)(y*Cell_X*N_BIN+x*N_BIN+k);
if(sum_magnitude == 0){
pHOGFeatures[num] = 0;
}else{
pHOGFeatures[num] = cell_hist[num] / sum_magnitude;
}
}
}
}
return 0;
}
/*
* 勾配強度と勾配方向を算出して HOG 特徴量を抽出
*/
int HOG(cv::Mat Image, double *pHOGFeatures, int Cell_Y, int Cell_X)
{
int num = Cell_Y*Cell_X*N_BIN;
double *cell_hist = new double[num](); // 0初期化が必要
CompHistogram(Image, cell_hist); // 勾配方向と勾配強度から勾配方向ヒストグラムを算出
CompHOG(pHOGFeatures, cell_hist, Cell_Y, Cell_X); //HOG 特徴量の算出
delete []cell_hist;
return 0;
}
int main(int argc, char** argv)
{
// 画像の読み込み
cv::Mat Image = cv::imread(argv[1], CV_LOAD_IMAGE_GRAYSCALE);
int Cell_Y = Image.rows / CELL_SIZE;
int Cell_X = Image.cols / CELL_SIZE;
int num = (int)Cell_Y*Cell_X*BLOCK_SIZE*BLOCK_SIZE*N_BIN;
double *HogVal = new double[num];
// HOG 特徴抽出
HOG(Image, HogVal, Cell_Y, Cell_X);
// 特徴量の書き出し
OutputHOGFeature(num, HogVal, argv[1]);
// 勾配画像の表示
Image *= 0.5; // 描画用のイメージを作成(輝度は半分にする)
for(int y=0; y<Cell_Y; y++){
for(int x=0; x<Cell_X; x++){
// 角度ごとに線を描画
cv::Point center(x*CELL_SIZE+CELL_SIZE/2, y*CELL_SIZE+CELL_SIZE/2);
for (int i = 0; i < N_BIN; i++) {
int num = y*Cell_X*N_BIN+x*N_BIN+i;
double theta = (i * THETA + 90.0 ) * PI / 180.0;
cv::Point rd(CELL_SIZE*0.5*cos(theta), CELL_SIZE*0.5*sin(theta));
cv::Point rp = center - rd;
cv::Point lp = center - -rd;
cv::line(Image, rp, lp, cv::Scalar(255*HogVal[num], 255, 255));
}
}
}
cv::imshow("Hog Image", Image);
cv::waitKey(0);
delete []HogVal;
return 0;
}まぁ、これで結果がこんな感じ。
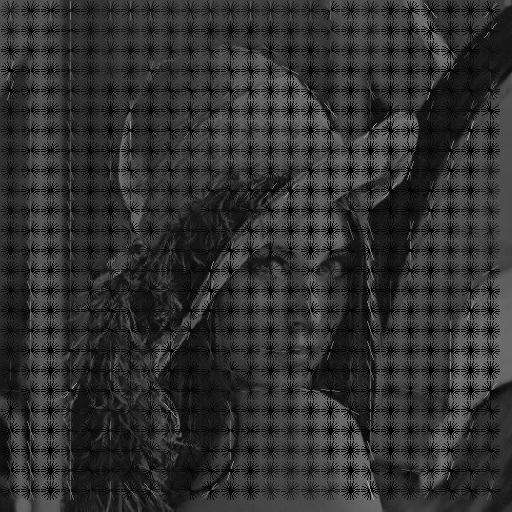
OpenCVの結果より色が薄く表示されているが、勾配の傾向は同じなのでこのまま使えそうな気配。 ちゃんと、特徴を抽出出来ていると思う。
Tweet write: 2016/06/22/ 23:24:40